近日,深圳大学物理与光电工程学院/生物医学光子学研究中心屈军乐教授团队在《Light: Science & Applications》(Nature子刊,影响因子17.782,中科院一区Top期刊)发表了题为《Fluorescence lifetime imaging for studying DNA compaction and gene activities》的研究论文。深圳大学Svitlana M. Levchenko副研究员为第一作者,屈军乐教授和美国纽约州立大学Paras N Prasad教授为共同通讯作者,合作单位包括波兰雅盖隆大学和美国纽约州立大学,深圳大学为第一完成单位。
目前,细胞核中基因组DNA折叠的调控过程是细胞生物学中的一个重要的基础问题,也是细胞生物学中研究最多但了解最少的重要问题之一。在活细胞环境中,研究细胞核中基因组DNA压缩以及生理过程或疾病发展过程中的动态重组极具挑战性。原因在于,要将巨大的基因组DNA包容至直径为5-10微米的微小的细胞核中,需要经过一个高度压缩的过程。此外,染色质的DNA压缩不是静态的,而是随着基因转录、翻译过程有时序变化。目前,光学显微技术是细胞生物学研究的重要工具,但传统光学显微成像技术有很大的局限性,即使是超分辨显微成像技术,其3D成像的分辨率也难以满足需求,因此限制了对复杂基因组结构的空间几何的研究,也无法追踪其动态转换过程。
屈军乐教授团队与美国纽约州立大学的研究团队合作,发展了一种用于表征DNA压缩动态过程的荧光寿命成像技术(FLIM),从而克服以往方法局限性。所发展的FLIM技术包括两种精确测量DNA压缩程度的方法:第一种方法基于加入DNA的荧光探针的荧光寿命与其局部微环境折射率之间的逆二次方关系,荧光探针的寿命随DNA压缩密度而变化,从而可表征DNA压缩程度;第二种方法是将荧光标记的核苷酸整合到DNA链中,通过一种荧光共振能量转移(FRET)技术获得其荧光寿命值的变化,从而反映出DNA的压缩程度的动态变化过程。研究结果证明,两种FLIM分析方法都可以成功表征DNA压缩的动态过程。
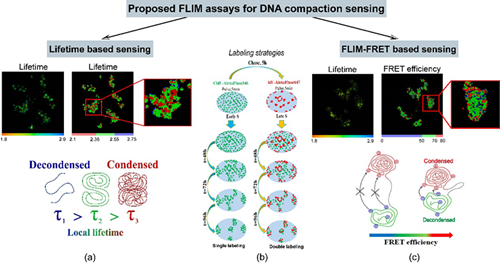
基于FLIM技术的DNA压缩研究方法示意图
该研究所发展的基于局域折射率和FRET的FLIM技术,能够定量绘制DNA压缩变化的图谱,为基因组结构组织的定量研究提供了新的光学表征手段,是揭示活细胞基因组发挥作用动态过程的有力工具,在细胞生物学机制研究中具有广阔的应用前景。
该研究得到了国家自然基金重点国际合作项目和深圳市科创委国际合作研究项目的资助。
论文信息:Svitlana M. Levchenko, Artem Pliss, Xiao Peng, Paras N. Prasad and Junle Qu, Fluorescence lifetime imaging for studying DNA compaction and gene activities. Light Sci Appl 10, 224 (2021).https://doi.org/10.1038/s41377-021-00664-w
论文地址:https://www.nature.com/articles/s41377-021-00664-w